PrimerStationは全ヒトゲノムに対して非常に高い特異性を持つ最適なプライマセットを計算するウェブサービスです。
本設計方法では高い精度を出すためにプライマの溶液中での会合率を用いました。DNA配列間のハイブリダイゼーション状態を会合率で厳格に計算するのは非常に計算時間がかかるため、これまでこのような物理化学モデルで計算し全ヒトゲノムに対してチェックをするような非常に特異的なプライマセットを表示するようなウェブサービスはありませんでした。
PrimerStationでは応答時間を短くするためにあらかじめ特異的なプライマの候補を100CPUの大規模な並列計算機(SunFire 15K)を用いて二ヶ月かけて計算しました。これによりPrimerStationは適格なプライマをインタラクティブに検索し出力することができます。
PrimerStationはプライマの交差反応を最小限にするような最適な温度範囲を求めることでマルチプレックスPCRに適した非常に特異的なプライマを選択することができます。またユーザはプライマ中のSNPをのぞくことやPCR産物中のAやCAのリピートを避けるといった経験則をプライマ設計に追加することができます。
私たちは無作為に選んだChrX上のターゲットに設計したプライマでPCR増幅を行い検証し、これらのプライマが目的のPCR産物を完璧に増幅していることを確認しました。この詳細は別途報告します。私たちはPrimerStationが多くの生物研究者と医学研究者の役に立つと考えています。
ゲノミックPCRプライマを設計するにはPCRのターゲットのRefSeq遺伝子IDもしくはゲノム領域を指定します。マルチプレックスゲノミックPCRプライマの設計では同時に増幅させるすべてのターゲットのゲノム領域を入力する必要があります。ターゲットとなるゲノム領域はカンマ、スペース、改行で区切って入力します。同時に増幅できるターゲット数は最大で40ゲノム領域に制限されています。
| 染色体領域 | chr染色体番号:開始位置-終了位置 (e.g., chr20:180000-190000) |
|---|---|
| ゲノム領域をターゲットとする場合上記のように記述します。染色体番号としては'chr1'-'chr22'、'chrX'、'chrY'が使用できます。ゲノムの開始位置から終了位置 までの範囲に増幅するターゲットが設計されます。 | |
| RefSeq遺伝子ID | 遺伝子アクセッション番号[@エクソン番号] (例 NM_003380@2) |
| RefSeq遺伝子ID遺伝子アクセッション番号と必要に応じてエクソン番号を指定します。 | |
|
NM_003380 chr20:180000-190000 chr12:2600000-2700000 |
この入力例では三つのゲノム領域(RefSeq遺伝子IDのNM_003380の領域、20番染色体上の180000から190000塩基、12番染色体の2600000から2700000塩基の領域)に対してマルチプレックスゲノミックPCRプライマを設計します。
|
NM_003380 NM_003380 NM_003380 chr20:180000-190000 chr20:180000-190000 |
この入力例ではNM_003380のmRNAの領域の異なる三カ所と20番染色体の180000から190000の領域の異なる二カ所の計五カ所にマルチプレックスゲノミックPCRプライマを設計します。
| NM_003380@1, NM_003380@3, NM_003380@8, |
この入力例ではNM_003380の一番、二番、八番エクソン上に計三つのプライマペアを設計します。この例のようにターゲットはカンマで区切ることもできます。
PrimerStationでは様々なオプションを指定して設計することができます。PCR産物長の範囲、PCR産物間の最小サイズ差、塩濃度、プライマ濃度、既知のSNPや二次構造やリピート配列を避けるかを指定することができます。
ゲノミックPCRプライマの設計ではPCR産物長の設定が非常に重要です。すべてのPCR産物を電気泳動によって同時に検出するために私たちはPCR産物の範囲を60から600塩基にし、これを設計の初期値としました。もしこの設定でプライマを設計できない場合には設計するPCR産物長の範囲を広げることでできるようになるかもしれません。
このパラメータは同じPCR産物長のマルチプレックスゲノミックPCRプライマを設計しないようにするためにあります。これは設計されたプライマのいくつかが同じPCR産物長を持つとそれらを大きさで区別することができないためです。PrimerStationでは簡単のため、PCR産物間の最小のサイズ差を指定することができ、PCR産物は互いに指定されたサイズ以上の差を持つようにプライマが設計されます。
SNP上に設計されたプライマはターゲット配列を完全には増幅することができません。PrimerStationは細心の注意を払ってこのようなハイブリダイゼーションの危険を防ぎます。チェックボックスをオンにすることで既知のSNP上にプライマを設計しないようにします。SNP情報はdbSNP(http://www.ncbi.nlm.nih.gov/projects/SNP/)を使用しています。
プライマ設計の失敗の原因の一つはプライマ配列の二次構造です。PrimerStationは望ましくない二次構造をもつようなプライマを設計しないようにします。プライマが取りうる最も安定な二次構造とその自由エネルギーをMfoldで計算します。安定な二次構造はこの自由エネルギーが小さくなります。セレクトボックスでこの自由エネルギーの最小値の閾値を指定することができます。この二次構造のチェックは計算に時間がかかります。
PCR産物中のポリA、ポリT、(CA)nもしくは(GT)nリピートはPCRスリップを引き起こし、PCR産物が意図しない大きさになってしまいます。このようなスリップを避けるためにはポリA、ポリT、(CA)nもしくは(GT)nリピートを含むようなPCR産物をプライマが持たないようにそれぞれ対応するチェックボックスをオンにする必要があります。
プライマ設計の結果は以下の図のようになります。
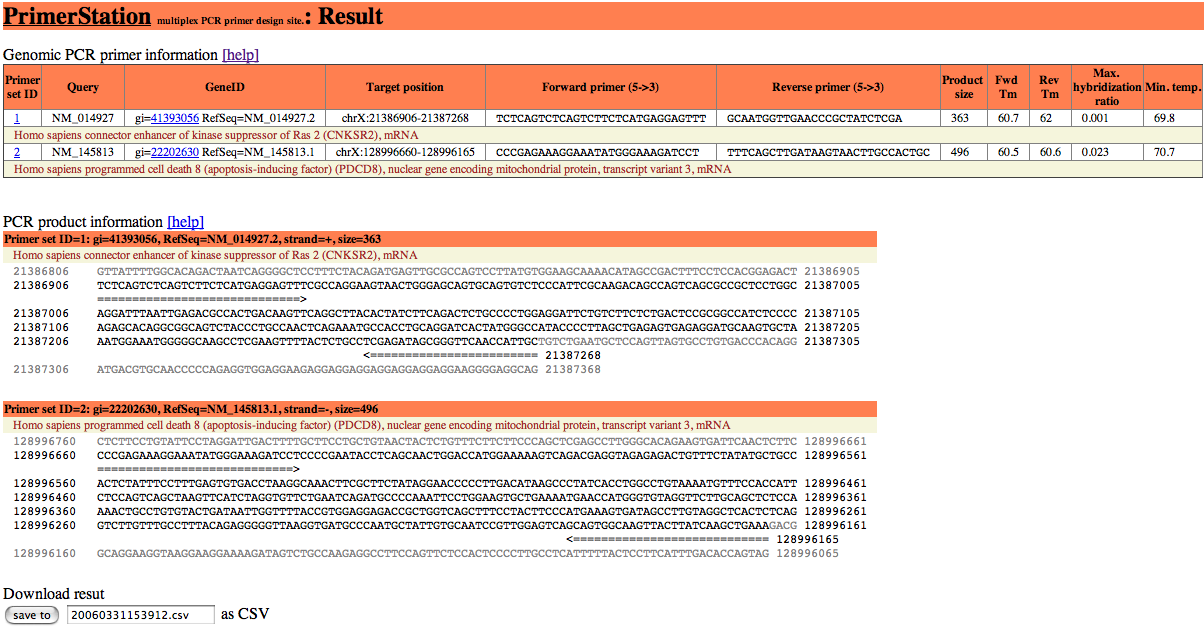
設計結果は二つの部分、設計されたプライマの情報とそのPCR産物の情報、からなります。設計結果はCSVファイルでダウンロードすることができます。
ここでは次のような情報から構成されています:プライマセットID、ユーザの入力、遺伝子ID、ターゲットの位置、フォワードプライマ配列、リバースプライマ配列、PCR産物長、フォワードプライマのTm値、リバースプライマのTm値、最大会合率の次に大きい会合率、最小実験可能温度。二次構造計算のオプションを指定した場合にはその結果も表示します。
プライマ情報の詳細は以下のようになります:
| Primer set ID | ターゲット配列の番号 |
|---|---|
| Query | ユーザの入力 |
| GeneID | 指定された遺伝子のGenBank ID (例:gi=41393056)とRefSeq ID (例:RefSeq=NM=014927.2) |
| Target position | 設計されたプライマのPCR産物のゲノム上の位置 |
| Forward primer | フォワードプライマ配列 |
| Reverse primer | リバースプライマ配列 |
| Product size | 設計され得たプライマのPCR産物長 |
| Fwd Tm | Nearest-neighbor法で計算したフォワードプライマのTm値 |
| Rev Tm | Nearest-neighbor法で計算したリバースプライマのTm値 |
| Max. hybridization ratio | プライマとオフターゲット候補との最大会合率 |
| Min. temp. | プライマの特異性が確保される最低アニール温度 |
ここではプライマで挟まれたPCR産物とその外側の配列を表示します。PCR産物は黒文字、PCR産物の外側は灰色文字で表示されています。プライマは矢印で示されています。
設計結果をCSV(comma separated values)形式でダウンロードできます。CSVファイルはマイクロソフトのエクセルで読み込むことができます。
PrimerStationへの全てのアクセスは記録され、アクセス、入力パターン、ヘビーユーザの統計を取るのに使用されます。ユーザによって提供された情報はPrimerStationの機能を強化するために使用されます。未解決のIPアドレスまたはプロクシサーバーからのアクセスは拒否することがあります。
本研究は文部科学省科研費特定領域研究 (C)と文部科学省リーディングプロジェクト「細胞・生体機能シミュレーションプロジェクト」の援助を受け、ソニー株式会社との共同研究により行ったものです。本研究のコンピューター計算時間は東京大学医科学研究所ヒトゲノムセンターのスーパーコンピュータシステムにより提供されました。